Foram anunciados pela academia real de ciências da Suécia, em Copenhague, Dinamarca, os ganhadores do prémio Nobel de Química 2017. Os cientistas Jacques Dubochet, Joachim Frank e Richard Henderson ganharam o prêmio por desenvolverem a cryo-electron microscopy (Criomicroscopia eletrônica), que determina estruturas tridimensionais de alta resolução de biomoléculas em soluções. Esse método levou a um grande avanço da bioquímica e ao entendimento básico da vida, além de contribuir para desenvolvimento de medicamentos e conceitos dentro da medicina e biomedicina. [1]
![Figura 1: Caricaturas dos ganhadores do prémio Nobel de Química 2017: Jacques Dubochet, Joachim Frank e Richard Henderson. [2]](http://www.petquimica.ufc.br/wp-content/uploads/2017/10/caricatura.jpg)
Figura 1: Caricaturas dos ganhadores do prémio Nobel de Química 2017: Jacques Dubochet, Joachim Frank e Richard Henderson. [2]
O trabalho laureado com o prêmio vem de uma pesquisa de muitos anos, para ser mais especifico, da década 70, quando Richard Henderson se propôs em determinar a estrutura de proteínas ativas. A principio, ele utilizou a difração de raios-X e a Ressonância Magnética Nuclear (RMN), no entanto as duas tecnologias apresentavam limitações quando aplicadas ao objetivo dele. A difração de raios-X exige que as moléculas formem cristais bem organizados, como os cristais de gelo, ou que os materiais sejam sólidos, sendo que essa forma rígida revela muito pouco sobre a dinâmica da proteína, e o RMN, apesar de conseguir determinar moléculas orgânicas em solução, não consegue fazer o mesmo para biomoléculas. Tais limitações fizeram com que o Henderson apostasse na Microscopia Eletrônica (ME), que funciona mais ou menos como microscopia comum, mas tendo um feixe de elétrons sendo enviado através da amostra, ao invés de luz. O comprimento de onda dos elétrons é muito mais curto do que o da luz, então o microscópio eletrônico pode tornar visíveis estruturas muito pequenas, até mesmo posições de átomos individuais. Em teoria, a resolução do microscópio teria sido mais do que suficiente para Henderson obter a estrutura molecular de uma proteína de membrana, mas na prática o projeto era quase impossível. [1]
Quando o microscópio eletrônico foi inventado, na década de 1930, os cientistas achavam que ele era adequado, apenas, para estudar matéria morta. O intenso feixe de elétrons, necessário para a obtenção de imagens de alta resolução, incinera o material biológico e, se o feixe for enfraquecido, a imagem perde seu contraste. Além disso, o microscópio eletrônico requer um vácuo no sistema, condição na qual as biomoléculas se deterioram, já que a água ao redor do material evapora. Quando as biomoléculas secam, elas colapsam e perdem sua estrutura natural, tornando as imagens inúteis. [1]
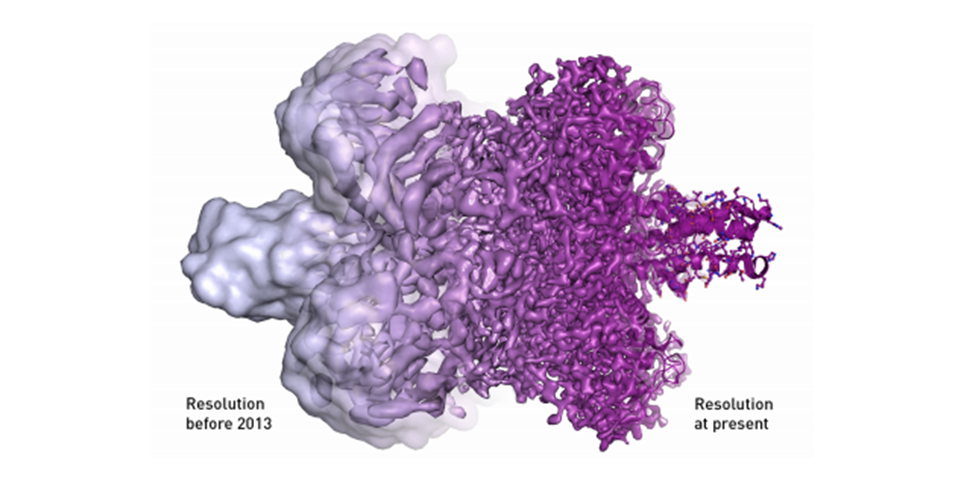
Mesmo com essas limitações Henderson não desistiu, e resolveu continuar apostando na ME. Utilizando uma proteína chamada Bacteriorrodopsina, que é encontrada na membrana de organismos fotossintetizantes, ele aplicou ME na própria membrana com a proteína, evitando assim a perda estrutural dessa. Além disso, usando uma solução de glicose sobre a membrana, evitando assim a desidratação da proteína, e sobrepondo as imagens obtidas, ele conseguiu obter a estrutura tridimensional dessa proteína (Figura 1), no entanto isso só foi possível, pois esta se embala de forma natural e regular na membrana. Poucas outras proteínas configuram-se espontaneamente dessa maneira. A questão era se o método poderia ser generalizado, isto é, se seria possível usar um microscópio eletrônicas para gerar imagens de alta resolução, em 3D(tridimensional), de proteínas, que foram espalhadas aleatoriamente na amostra e orientadas em direções diferentes. [1]
Outro laureado com o prêmio, Joachim Frank, conseguiu responder essa questão olhando de um ponto de vista computacional. Utilizando novamente o ME, ele obteve imagens 2D (bidimensionais) com baixa resolução de um ribossomo em uma forma aleatória. Unindo essas imagens em um algoritmo ,que ele e sua equipe desenvolveram, foi possível obter uma imagem 3D de alta resolução da proteína. [1]
Embora, no trabalho de Henderson com a proteína Bacteriorrodopsina, a utilização da solução da Glicose para evitar a desidratação tenha dado certo, em outros sistemas essa estratégia não seria possível. No entanto, Jackes Debuchet conseguiu resolver esse problema: a princípio, ele pensou em evitar a perda de água congelando o sistema, mas os cristais de gelo interferiam na passagem dos feixes de elétrons, o que tornava o processo inviável. Após isto, ele utilizou o método de vitrificarão, que consiste em congelar a água de forma rápida, com etano líquido na temperatura de -190°C, criando um estado amorfo da água que possibilitava a passagem do feixe de elétrons. Para testar a eficiência do seu método, ele o aplicou na obtenção de estrutura cristalina de um vírus, na qual obteve êxito. [1]
Juntando a contribuições de todos esses pesquisadores, foi possível a construção desta tecnologia que contribui de diversas formas para ciência, seja na bioquímica, na medicina, ou até mesmo na biomedicina. Um fato que marca essa contribuição foi aplicação da Crio-ME na determinação da estrutura tridimensional do vírus da ZIKA. Durante o surto que ocorreu recentemente dessa Doença, que estava causando a microcefalia nos bebês recém-nascidos, foi possível, com essa determinação tridimensional, conhecer o mecanismo de ação do vírus e o começo da busca de uma vacina. [1]
Leia mais em: https://www.nobelprize.org/nobel_prizes/chemistry/laureates/2017/popular-chemistryprize2017.pdf
[1] POPULAR SCIENCE BACKGROUND, They captured life in atomic detail; Disponível em: https://www.nobelprize.org/nobel_prizes/chemistry/laureates/2017/popular-chemistryprize2017.pdf,; Acesso:15/10/2017
[2] Nobel Media. Ill. N. Elmehed, The Nobel Prize in Chemistry 2017; Disponível em: https://www.nobelprize.org/nobel_prizes/chemistry/laureates/2017/ ,; Acesso: 15/10/2017
![Figura 2: Estrutura Tridimensional da Bactetiorrodopsina. [1]](http://www.petquimica.ufc.br/wp-content/uploads/2017/10/bact.png)
![Figura 3: Esquema do processo e do desenvolvimento da analise computacional de Frank. [1]](http://www.petquimica.ufc.br/wp-content/uploads/2017/10/er4defde4rf.png)
![Figura 4: Estrutura tridimensional do Vírus da ZIKA. [1]](http://www.petquimica.ufc.br/wp-content/uploads/2017/10/zica.png)